|
|
Introduction
SPLASH
Summary
SPLASH(sequencing of psoralen crosslined, /igated, and selected hybrids)可在细胞内(in vivo)捕获全基因组(global-scale)水平上的RNA分子内及分子间的互作。
SPLASH Strategy and Specificity
Biopsoralen(生物素标记的补骨脂素)作为交联剂,在365 nm UV光照射条件下,与细胞内互作的碱基对片段交联在一起(Psoralen crosslinking)。交联的RNA被从细胞中提取出来并片段化,通过streptavidin beads(链霉亲和素珠)富集,然后进行邻近绑扎(proximity ligation)、逆交联(reverse crosslinking)、引物连接、反转录、环化PCR扩增后生成测序文库(图1.1)。文章中还提到如何提高biopsoralen进入细胞的比例,交联的可逆性(reversibility)对于实验的必要性,选取环化扩增(circularization protocol)而不是传统线性扩增以提升交联片段富集程度及结果精度,最后在大肠杆菌与酵母中验证了SPLASH可以检测先前已知的RNA-RNA互作。这里我们只关注基本原理及分析结果,这部分就不再赘述。
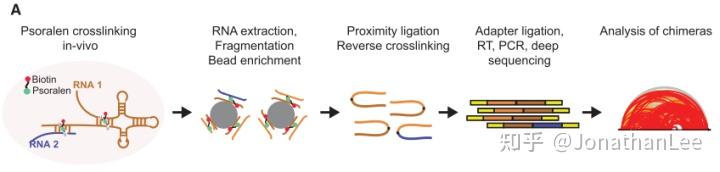
图1.1 SPLASH的实验设计示意图
文库产生Fastq文件后,文章通过Split-Read Mapping流程鉴定interaction,并进行一系列质控,具体来说:
- 使用SeqPrep移除adapter(引物序列)
- 使用BWA MEM进行Split-Read Mapping,chimeras的两个来源的reads会被分别比对到两个位置
- 只保留split read > 50bp的chimeras
- PCR duplicate removal
- 只保留unique mapped chimeras
- 移除潜在的splicing junction处的chimeras
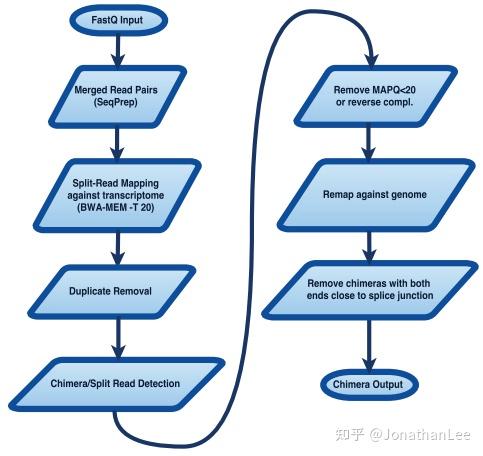
图1.2 SPLASH文库分析流程图
文章利用一系列指标证明SPLASH鉴定的RNA interactions具有高敏感、高特异性,且与其它类似技术相比,在检测分子内部互作上与其他技术结果相当,在检测分子间互作上,效果显著提升。具体来说:
- SPLASH预测结果与实验解析的80S核糖体复合物结构很好的吻合
- 将人与酵母的转录本混合以测定随机互作的比例,即假阳性率(false discovery rate),结果发现FDR<3.7%
- 通过一组对照试验证明biopsoralen交联对于保持RNA互作的特异性至关重要
Main Results
1. Global Structure of the Yeast and Human RNA Interactomes
作者在四个人类细胞系(HeLa cells,类淋巴母细胞(lymphoblastoid cells),胚胎干细胞(ESCs)及视黄酸分化细胞(retinoic acid differentiated cells))及两种酵母细胞中进行SPLASH。这里我们只关注于四种人类细胞系中得到的结果。最终捕获来自1,311个基因的3,497个分子内mRNA互作(intramolecular mRNA interactions)及84个分子内lncRNA互作。分子间相互作用非常多样普遍(图1.3)我们这里只关注RNA-RNA interactions,后续关于RNA分子内互作便不再介绍。
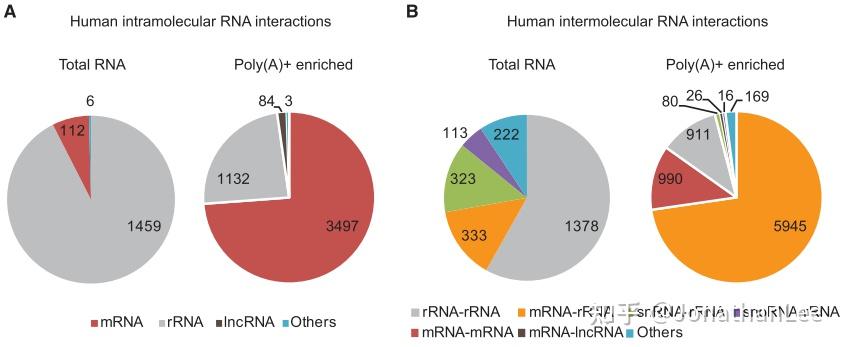
图1.3 人类细胞系中RNA互作结果概览
2. SPLASH Uncovers New rRNA-rRNA and snoRNA-RNA Interaction Sites
鉴定snoRNA-rRNA互作,尤其是H/ACA snoRNA的互作,是非常有挑战性的。SPLASH不仅鉴定得到先前已知的RNA-RNA互作,包括5.8S-28S核糖体RNA互作、U4-U6 snRNA互作和U2-U6 snRNA互作,也鉴定得到211对snoRNA-rRNA互作(78个snoRNA,55个C/D box和23个H/ACA snoRNA),其中122对是先前未发现的。
3. mRNA-mRNA Interactions Define Modules of Coregulated Genes
有趣的是,SPLASH鉴定得到近1,000条mRNA-mRNA互作,且大多数互作都发生在转录本相同两端(一条mRNA的5&#39;UTR与另一条mRNA的5&#39;UTR互作,或一条mRNA的3&#39;UTR与另一条mRNA的3&#39;UTR互作),同时也有相当一部分发生在一条转录本两端与另一条转录本更远的位置互作(一条mRNA的5&#39;UTR与另一条mRNA的CDS区域互作)(图1.4)
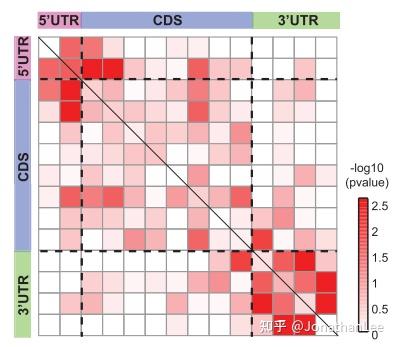
图1.4
LIGR-seq
Summary
LIGR-seq(LIGation of RNA-RNA duplexes crosslinked in vivo)可在细胞内捕获全基因组水平上RNA-RNA互作。
LIGR-seq Strategy and Specificity
LIGR-seq的实验方法与SPLASH类似。使用AMT一种psoralen的衍生物作为交联剂,在365 nm UV光照射条件下,与细胞内互作的碱基对片段交联在一起,并在并列的尿嘧啶间添加加合物(adducts)。然后裂解细胞,移除rRNA, 使用限制性S1内切酶(limited single-strand S1 endonuclease)降解单链RNA。交联片段通过环形连接酶(circRNA ligase)连接成环状,然后再通过RNase R(一种 3^{&#39;} \rightarrow 5^{&#39;} 核酸外切酶)消化未交联的RNA,从而达到富集的效果。然后进行逆交联,构建测序文库。(图1.5)
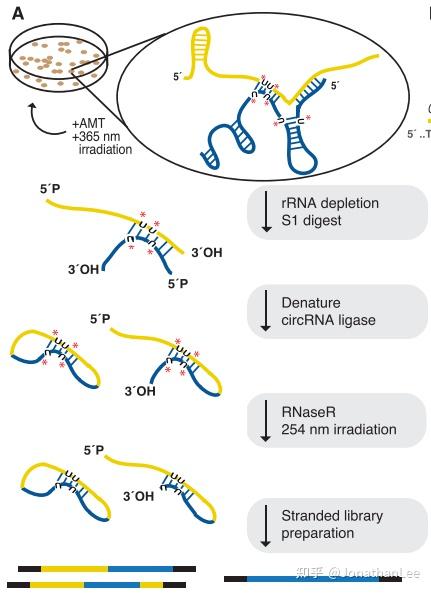
图1.5 LIGR-seq的实验设计示意图
LIGR-seq的数据处理流程与SPLASH也很类似,但细节上更讨巧,进行了更严格的质控。进一步,以+AMT/-AMT group用统计学模型做校正很大程度上移除了部分因为RNA随机结合产生的假阳性结果。具体来说:
- 计算两个互作基因之间的联合概率 P(g_x,g_y) ,联合密度函数为: \begin{eqnarray} pdf(g_x,g_y) \varpropto \left \{ \begin{array}{rcl} 2P(g_x)P(g_y),if \ g_x:g_y is \ observed \ and \ g_x \neq g_y \\ 0 ,else \ g_x:g_y \ is \ \urcorner \ observed \ or \ g_x = g_y\end{array} \right. \end{eqnarray} \\ 其中 P(g_x) = \frac{g_xRPM}{\mathop{\sum}\limits_{i=1}g_iRPM} \\ 我们标准化概率分布至和为1: pdf(g_x,g_y) = \frac{P(g_x,g_y)}{\mathop{\sum}\limits_{i=1}\mathop{\sum}\limits_{j=i+1}P(g_i,g_j)} \\ g_x:g_y interaction的总counts数 k 服从分布 k \sim Binomial(pdf(g_x,g_y),N) ,然后计算显著性p-value以及进行Bonferroni校正。
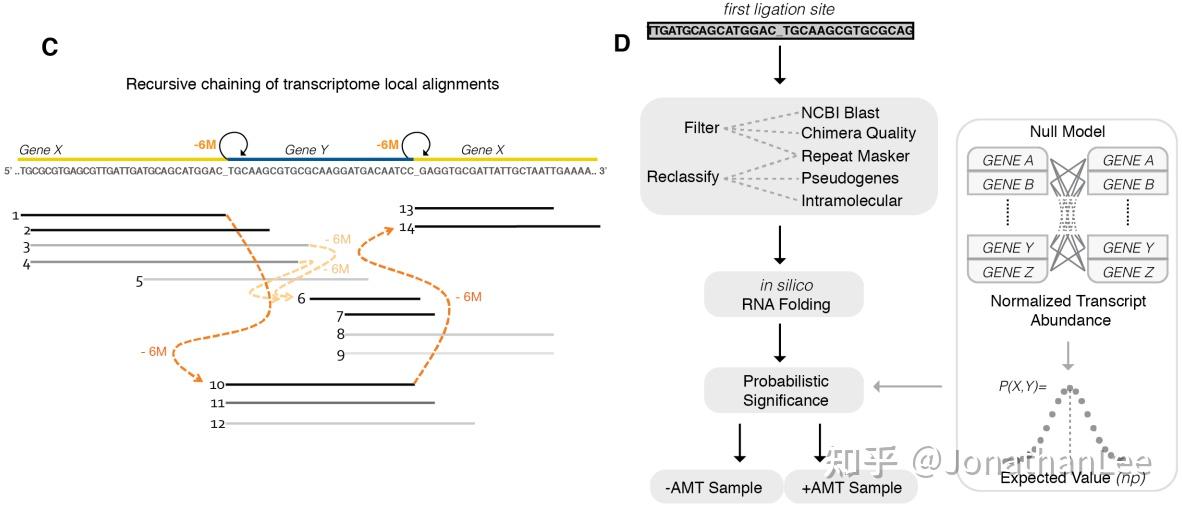
图1.6 LIGR-seq文库分析流程
Main Results
1. Global Snapshot of the RNA-RNA Interactome
LIGR-seq总共鉴定得到514对显著的interactions,大部分都是rRNA-rRNA与snRNA-snRNA互作。
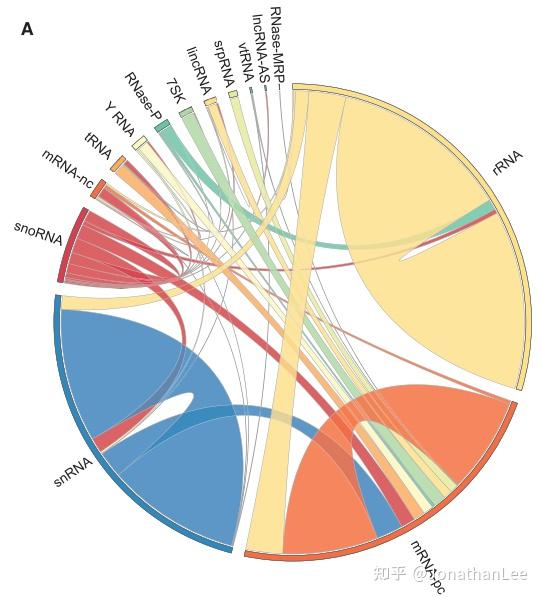
图1.7 LIGR-seq鉴定的RNA-RNA互作景观
2. SNORD83B Controls the Levels of Target mRNAs
PARIS
Summary
PARIS Strategy and Validation
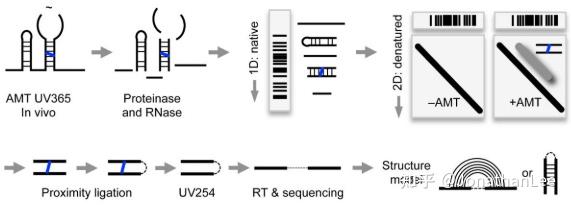
图1.8 PARIS的实验设计示意图
PARIS分析流程与上述技术类似,包括引物移除、PCR duplicates移除、多比对以及non-gapped reads移除、spliced reads移除。
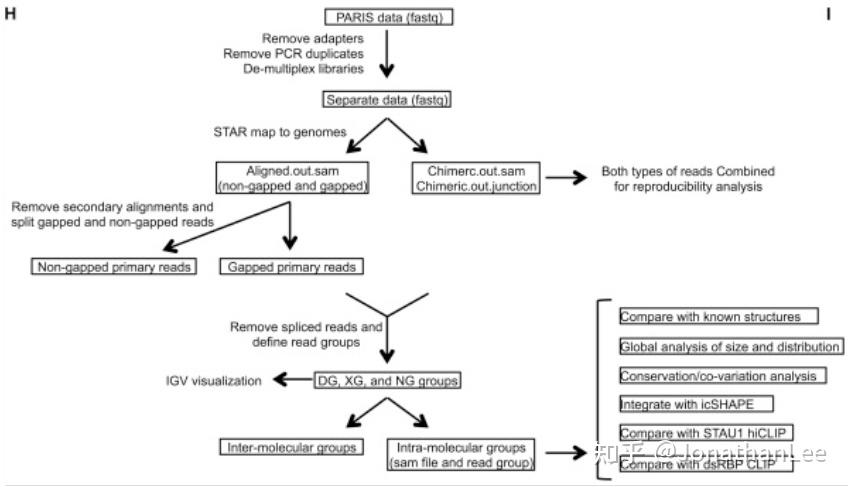
Main Results
PARIS主要关注RNA的二级结构信息,做出了许多详尽漂亮的结果,其中许多分析结果都值得借鉴。对于RNA的long-range structure,alternative structure以及互作区域的phylogenetic feature进行了详细阐述。但只有一个result关注RNA-RNA interaction,介绍了PARIS鉴定出的一些新的互作关系,我们在本文中只关注RNA-RNA interaction,就不展开详细介绍。
RIC-seq
Summary
RIC-seq(RNA in situ conformation sequencing)与前面三种方法在实验原理上有所区别,RIC-seq是在细胞内全局捕获由RNA结合蛋白(RNA binding protein)介导的RNA分子内及分子间互作。
RIC-seq Strategy and Validation
RIC-seq使用formaldehyde(甲醛)交联,用micrococcal nuclease(MNase)随机切割RNA并在切割后游离的3&#39;端去磷酸化,暴露羟基基团。RNA 3&#39;端用pCp-biotin标记(biotinylated cytidine phosphate)随后用FastAP alkaline磷酸酶移除pCp-biotin末端磷酸基团并用PNK(T4 polynucleotide kinase)添加磷酸基团,然后进行proximity ligated。随后便是常规的纯化、片段化、streptavidin beads富集以及文库构建。
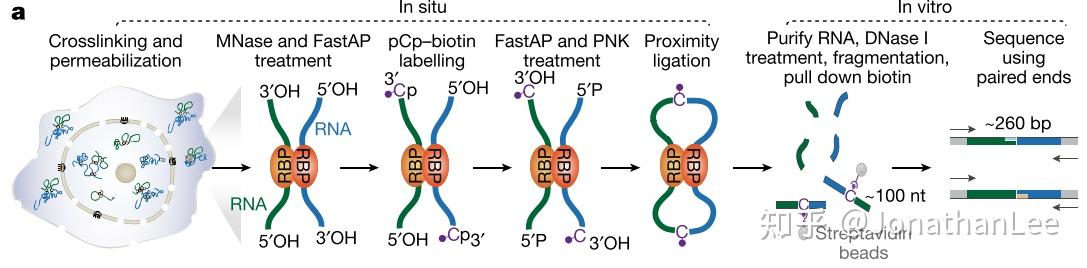
图1.9 RIC-seq 的实验设计示意图
RIC-seq的处理流程与其他技术的处理流程类似,包括去接头、去PCR重复、去低重复度序列、chimeric序列分割后分别比对、移除splicing junction位点处的reads等。不同的地方在于rRNA只用于验证技术的可靠性,没有关注rRNA与其他类型RNA的互作关系(图1.10)。在鉴定高可信度的intramolecular interaction以及intermolecular interaction层面,文章分别用了两种已发表的模型进行统计学检验。具体来说:
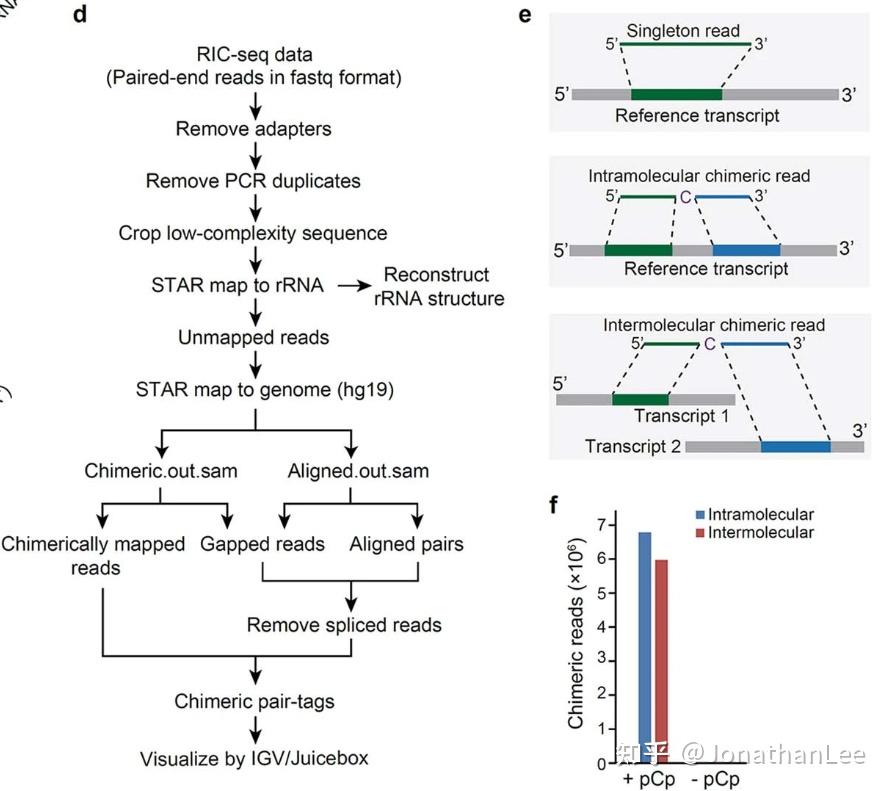
图1.10 RIC-seq的实验设计示意图
经验证,RIC-seq很好的捕获到rRNA的三级结构,与冷冻电镜得到的28S rRNA结构相比较,经ROC验证,精度可达89%,与PARIS结果相当。此外,RIC-seq也很好的捕获到lncRNA的3D结构(7SK,SRP以及RPPH1),并且与PARIS结果相当。
Main Results
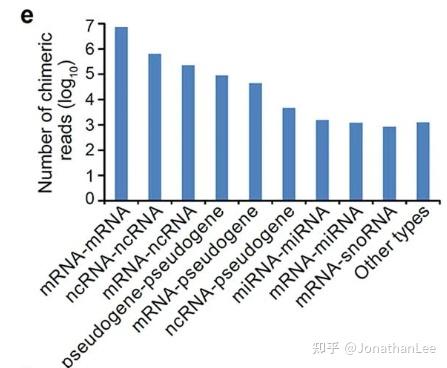
- Global View of RNA-RNA Interactions
RIC-seq捕获近百万条RNA-RNA interactions,其中大部分是mRNA-mRNA interactions,ncRNA-ncRNA interactions以及mRNA-ncRNA interactions。
其中有些lncRNA展现出广泛的trans interactions(反式相互作用)。这些lncRNA倾向于结合高表达的转录本,更有趣的是,这些lncRNA甚至即结合某些RNA转录本,又结合这些RNA的DNA loci。此外,文章还发现NEAT1与MALAT1存在紧密的相互作用。
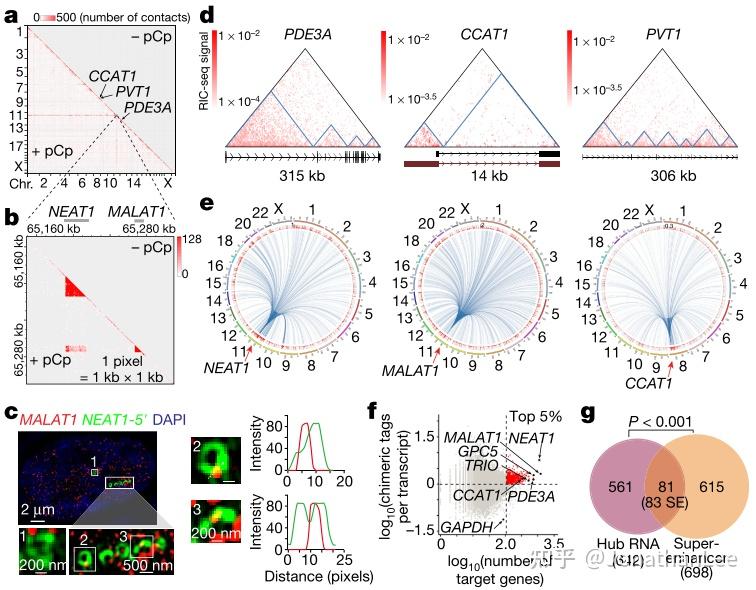
2. Features of RNA spatial interaction
3. Enhancer-promoter RNA interaction map |
|